Не африканцы - мы! / начало
Ещё из исследований и дискуссий по генетике и материалы с Клёсовым
«Выход из Африки»: те же грабли, только в разрезе
Попгенетика и ДНК-генеалогия
К вынесенной в заглавие теме приходится возвращаться снова и снова, но мне не в тягость. Во-первых, тема интересна с точки зрения методологии современной науки, в данном случае популяционной генетики, перенесенной на почву исследования генома. Техническая сложность резко повысилась, но попгенетики остались теми же, при вопиющем отсутствии у них адекватной научной школы. ©По теме: Невероятные неандертальцы

Neandertal Museum in Mettmann, Germany
Во-вторых, на тему геномных исследований практически нет научно-популярных статей, отчасти потому, что популяционные генетики обыкновенно косноязычны, статьи они пишут по стилю и способу изложения ужасные, и научно-популярные статьи у них получаются как на том заводе, который что ни делает, а получается танк. Иначе говоря, у них получается не научно-популярная статья, а такое же вязкое, неудобоваримое «изложение».
Наконец, они не могут писать доходчивых научно-популярных статей по результатам изучения генома человека, потому что они сами не понимают смысл того, что делают. Они берут сотни тысяч и миллионы геномных снипов, что-то там складывают и вычитают по неким прописям, удаляют то, что им по разным причинам не нравится, причем удаления достигают до трети исходных снипов, потом закладывают в компьютер, тот пережевывает и что-то выплевывает, они это «интерпретируют», причем так, что волосы дыбом становятся, и результат публикуют как якобы научное исследование.
Но это еще полбеды, а самая беда у попгенетиков от генома имеет ментально фундаментальный характер. Главный критерий у них - «похожесть». Вот эту «похожесть» они оформляют в самых разных видах. Основной - это «метод главных компонент». Вся его суть - это найти «похожесть» между сравниваемыми величинами, которые берутся самые разные. Если немного утрировать, но только немного - они берут геномы некого индивидуума и его дяди, то есть брата отца. Геномы оказываются похожими, что неудивительно. Незамедлительно следует вывод, что этот индивидуум произошел напрямую от дяди. Или, напротив, дядя - от индивидуума. Это уже как они порешат. Это и называется «интерпретация». Примеров в академических журналах - масса…
Только недавно мы разбирали статью, в которой нашли, что геном ископаемых ямников (это - древние обитатели ямной культуры) похожи на геном древней культуры шнуровой керамики. Вывод - люди шнуровой керамики произошли от ямников, и получили от последних индоевропейские языки. То, что у ямников гаплогруппа R1b, а в шнуровой керамике - гаплогруппа R1a, на это они внимания не обратили. Гаплогрупп в статье они вообще не разбирали. Видимо, посчитали лишним - геном же похож, не так ли? То, что R1a и R1b - это «братья», которые произошли от одного «родителя» - гаплогруппы R1, и поэтому геном у них обязан быть похож - об этом, видимо, не подумали. На гаплотипы и не посмотрели, да их и не определяли. А гаплотипы сразу бы показали, что между R1a и R1b - не менее 40 тысяч лет разницы, да и между гаплотипами ямников и европейцев, древних или современных - тоже тысячелетия разницы. Не могли носители гаплогруппы R1b, эрбины, пройти напрямую из ямной культуры в Европу, во всяком случае, те ямники, геномы которых изучали.
Наконец, те субклады гаплогруппы R1b, которые были у тех ямников, а именно в основном R1b-L23-Z2103, в современной Европе почти не наблюдаются, их нашли у всего нескольких европейцев из десятков тысяч носителей гаплогруппы R1b в Европе, причем почти все европейцы имеют субклады R1b-U106 и R1b-P312 и нисходящие от них, которые не произошли от ямных R1b-L23-Z2103, это совсем другие ДНК-линии. Иначе говоря, не прошли те ямники и их потомки в Европу. Они, в лучшем случае, прошли на юг, на Кавказ и в Месопотамию, а в худшем (для них) случае - просто вымерли. Так что не принесли они индоевропейские языки в Европу, да и не могли принести. И эта грубая ошибка геномных попгенетиков произошла только потому, что не знают они ДНК-генеалогии, не хотят рассматривать.
Это просто один пример из десятков, когда «похожесть» геномов прямолинейно и бестолково берется как главный критерий. Ситуация напоминает ту, которая была в археологии в середине и второй половине прошлого, 20-го века, когда такой же критерий «похожести» показал свою несостоятельность, и в итоге археологи надолго отказались от изучения древних миграций, когда изучение основывалось на «похожести». Они поняли, что не могут отделить миграции от «культурной диффузии», и не могут определить направления миграций, когда основывались на «похожести» - то ли отсюда туда, то ли оттуда сюда. Тогда же был выдвинут лозунг «горшки - не люди», в том смысле, что похожие горшки в двух разных отдаленных местах не означает, что создатели этих горшков передвигались вслед за ними. Так что пора геномным попгенетикам, наконец, сообразить, что похожесть геномов не указывает на направления миграции древних людей, и перестать наступать на те же грабли.
Технически современная генетика вышла на такие рубежи, о которых нельзя было и помыслить еще несколько лет назад. Стало возможным выявлять в ДНК любого человека миллионы снипов, то есть практически необратимых мутаций, которые составляют уникальную мозаику, то есть картину снипов каждого. Эта мозаика отражает миллионы лет эволюции человека, и в эту картину входит множество снипов, унаследованных от общих предков человека и древних предков макаки, орангутанга, гориллы, шимпанзе. Эти снипы затем ушли к неандертальцам (у нас с неандертальцем общих снипов не менее 99%), древним африканцам, древним неафриканцам и их потомкам, и они есть в ДНК любого из нас, миллионы этих древних снипов.
Ну, и что с этим добром делать? Техника - в буквальном смысле на грани фантастики - нам это предоставила, и, как это бывает, обогнала мыслительный аппарат человека. Мыслительный аппарат эти миллионы снипов не схватывает, вручную их обработать нельзя. К счастью, компьютерная обработка данных тоже вышла на уровень граней фантастики, как и способы хранения и переработки информации. Осталось все это совместить.
И вот здесь начинаются большие проблемы. Человек ведь мыслит линейно, за редким исключением людей очень одаренных, которых называют гениями. Поэтому ординарные люди, исследователи генома, стали, естественно, применять к столь сложной проблеме простые линейные подходы, рассуждения, размышления. Выявил компьютер, что у американских индейцев усредненный геном на 11% схож с геномом усредненного русского, вот исследователь и записал: (1) индейцы с русскими совпадают по геному на 11%, значит, (2) русские произошли от американских индейцев. Причем, заметим, что это не компьютер говорит, это исследователь так трактует данные. Как он, исследователь, захочет. Причем, заметьте, не американских индейцев выводит из русских, а наоборот, хотя 11% похожести можно трактовать в любую сторону. На самом деле там похожесть вообще не имеет отношения к поставленному вопросу. У самого исследователя геном на 95+% схож с геномом шимпанзе, видимо, можно с таким же успехом записать, что шимпанзе произошел от того же исследователя. Разберем это чуть подробнее, нам это понадобится для дальнейшего изложения.
История с шимпанзе
Начнем с того, что компьютер сам не скажет, что геномы русского и американского индейца совпадают на 11%, или на сколько там еще. Для этого исследователь закладывает в компьютер программу и исходные условия, определения и допущения. Что там «совпало на 11%»? Там ведь миллионы снипов, включая снипы от общих предков с шимпанзе и прочих приматов, и совпадение с ними уже больше, чем на 90%. Иначе говоря, 11% - это уже означает, что большинство снипов «отфильтрованы», причем фильтрование тоже производится на условиях и по правилам, закладываемых исследователями. Это они диктуют компьютеру, как надо сортировать и фильтровать данные. При этом надо в первую очередь отфильтровать общие снипы древнего общего предка человека и шимпанзе. А его генома у нас нет. Поэтому генетики берут его «прокси», то есть представителя-заместителя, а именно, геном шимпанзе из современного зоопарка. Но наш шимпанзе - неважный прокси, так как его отделяет от того общего предка около пяти миллионов лет. За это время новых снипов у нашего шимпанзе образовалась масса, в абсолютном исчислении. Можно даже подсчитать, сколько. При скорости мутации 0.7х10-9 на пару нуклеоидных оснований в год, за 5 миллионов лет набежит в Y-хромосоме с ее 58 миллионов пар оснований 0.7х10-9 х 5х106 х 58х106 = 203 тысячи мутаций.
Часто работают с фрагментом Y-хромосомы размером в 10 миллионов пар оснований, но и там набежит 35 тысяч «лишних» мутаций, которые есть у нашего шимпанзе, но которых не было 5 миллионов лет назад у его общего с человеком предка. Иначе говоря, когда «фильтруют» снипы, то вычитают 203 тысячи лишних мутаций в расчете на Y-хромосому. Но какие лишние - неизвестно, потому что исходных данных давностью 5 миллионов лет назад нет.
Хуже то, что и шимпанзе разные, так что с мутациями опять неясность. Вот - простой пример. Работая над публикацией про гаплотипы древнего предка человека и шимпанзе, мы с коллегами провели поиск в геномной базе данных (European Nucleotide Archive и National Center for Biotechnology Information (NCBI) GenBank) по основным маркерам шимпанзе из панели наиболее «медленных» маркеров Y-хромосомы, принятых для человека, и получили следующие аллели, то есть в данном случае количество тандемных повторов (DYS 426, 388, 392, 455, 438, 578, 641, 472, 425, 594, 436, 490, 617, 568, 640, 492).
8 15 10 4 5 9 10 5 10 4 4 7 4 4 8 9
Это и есть гаплотип современного шимпанзе, во всяком случае, того, геном которого был заложен в базы данных, указанные выше. Как показали расчеты по этим маркерам, общий предок современного человека и шимпанзе жил 4.0±1.4 миллионов лет назад, что совершенно разумно, и согласуется с антропологическими датировками.
Через некоторое время в базе данных YSearch под номером 6RCUU появился гаплотип шимпанзе, представленный туда известным генетиком Томасом Краном, из источника, который описан так: Chimpanzee cell line Coriell NA03448. Гаплотип оказался совсем другим, хотя аллель при одном маркере - DYS641 - совпадала, была равна 10. Маркеры в основном тоже были другими (DYS393, 426, 388, 389-1, 458, 437, GATA H4, 570, 442, 578, 537, 641, 511, 444, 520, 446, 640, 492, 461, GATA A10, GAAT1B07, 441, 485, 533, 717), причем удивительно, что Кран использовал столь «быстрые» маркеры, ведь применять их к шимпанзе большого смысла не имеет, если сравнивать с маркерами человека. Впрочем, возможно, что у Крана были другие задачи. Но сейчас для нас главное не задачи Крана, а то, что на медленных маркерах общий предок современного человека и шимпанзе (по данным Крана) жил 3.8±1.5 миллионов лет назад, что тоже вполне разумно. Иначе говоря, шимпанзе Крана, как и «наш» шимпанзе, вполне «легитимны», но совершенно различны по гаплотипам. Гаплотип шимпанзе Крана был следующий:
10 10 12 12 15 14 9 4 14 5 9 10 13 10 18 21 12 10 13 5 7 9 14 9 19
и на самых медленных маркерах был совсем другой по сравнению с «нашим» шимпанзе:
10 12 5 10 12 10 (шимпанзе Крана)
8 15 9 10 8 9 (наш шимпанзе)
(маркеры DYS 426, 388, 578, 641, 640, 492).
И вот теперь главный вопрос - геном какого шимпанзе вычитали геномные попгенетики? Проверяли ли они на вычитание другого шимпанзе? Зная попгенетиков, ответ совершенно ясен - конечно, не проверяли. Как получилось, так и получилось. Но тогда какую научную ценность имеет то, что осталось после вычитания? Вопрос, впрочем, риторический. Кого бы там ни вычитали, все равно вычитали не тот набор мутаций, и в остатке после вычитания оказываются как сотни тысяч лишних снипов (которых не оказалось в данном конкретном шимпанзе в зоопарке), так и сотни тысяч снипов недостающих (которые были избыточно вычтены).
И вот сравнивают геномные попгенетики этот перекошенный геном после избыточного или недостаточного вычитания, скажем, с геномом неандертальца (точнее, неандерталки, все расшифрованные геномы неандертальцев, во всяком случае до последнего времени, принадлежали самкам), и говорят - о, смотрите, в них есть мутации, которых не было у шимпанзе (у того шимпанзе, из зоопарка!), но есть у современных людей. Значит, в нас есть доля неандертальцев! Мы произошли от неандертальцев! Пресса, конечно, сходит с ума от экстаза, бросает в воздух чепчики, не понимая, и не желая понимать и задавать вопросы (а зачем это им - сенсация ведь), что это не доля неандертальца, а просто не то вычитали. Из генома неандертальца ведь опять же того шимпанзе вычитали, из зоопарка, и опять сальдо-бульдо получилось дефектным. Причем поскольку тот, из зоопарка, был тем же, «референсным», то недостача или избыток снипов как у человека, так и у неандертальца были симметричными, одними и теми же. Вот и оказывается, что у человека тот же избыток снипов, что и у неандертальца. Сколько? Да те же 3%, которые находят по всему миру, кроме африканцев. Поскольку они разошлись с неафриканцами примерно 160 тысяч лет назад, то у них в определенной степени набежали одни снипы и не набежали другие, вот и разница.
И тогда станет понятно, почему исследования Крингса в 1997 году, которые тогда считали очень важными, а теперь и не упоминают, и которые показали, что мтДНК у современных людей и у неандертальцев принципиально расходятся (см. рис. ниже)
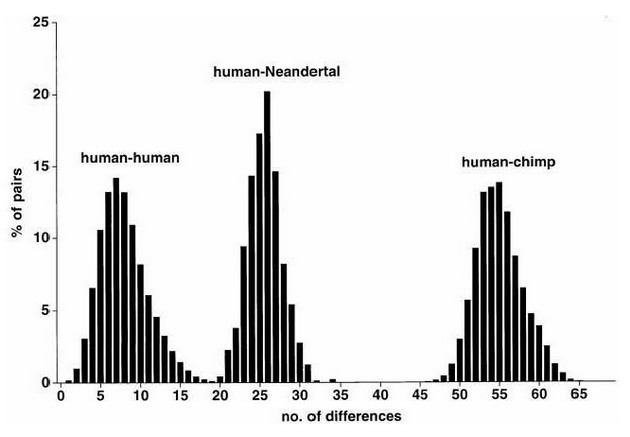
Среднее различие в последовательностях мтДНК между различными людьми на планете (первая диаграмма, среднее различие в 8±3 нуклеотидов), между людьми и неандертальцами (вторая диаграмма, среднее различие в 27±2 нуклеотидов), и людей и шимпанзе (третья диаграмма, среднее различие в 55 нуклеотидов). Такие же результаты были найдены и с другим неандертальцем, найденным в 2500 километрах от первого.
Вот так и получается - по мтДНК никакого близкого сходства между современными людьми и неандертальцами нет, по Y-ДНК не изучали (потому что все изученные по ДНК неандертальцы, повторяю, самки), но невероятно, что гаплотипы современных людей будут похожи на неандертальские, а геном при всех упомянутых манипуляциях оказался схож на 3%. И никто не приводит погрешности этих трех процентов, школа такая у попгенетиков, точнее, ее отсутствие. Что, если там 3±15%, что, скорее всего, и есть?
А то, что у африканцев этих трех процентов нет, как уверяют нас геномные исследователи, в то время как у неафриканцев есть, как-то ломает «теорию выхода» всех людей из Африки. Потому что тогда у всех людей, черных и белых, должны быть те самые 3% генома неандертальцев, куда же он делся? Остается вспомнить известный анекдот про трусы и крестик. Правда, и там нашли какое-то неуклюжее объяснение, не будут же попгенетики из-за каких-то неандертальцев терять столь ценную теорию выхода из Африки…
Конечно, я излагаю здесь картину в качественном виде, чтобы показать, в чем фундаментальные проблемы у попгенетиков с геномным анализом. Они сейчас, конечно, начнут выискивать у меня несущественные мелочи, типа что их «референсный» шимпанзе был не из зоопарка, а из Национального парка, только это сути не меняет. У него в любом случае около пяти миллионов эволюционной разницы с тем, геном кого надо было вычитать. Другие скажут - критиковать легко, а что вы сами предлагаете? Я отвечу, что предлагаю честно рассматривать и описывать данные в научных публикациях, и честно же сообщать, где данные шаткие или просто неверные, а не делать вид, что все правильно и других вариантов нет. Далее, я предлагаю пересчитывать данные не с одним шимпанзе, а с несколькими, поскольку геном получен не только для одного. Тогда станет ясно, что «доля неандертальца» в нас не те 3%, о которых постоянно докладывают, а может оказаться просто нулевой. И тогда надо честно же признать, что ошибка вышла, не подумали, статьи придется отзывать. Да, неприятно, зато честно по отношению к науке и читателям, которые в конечном итоге те геномные исследования и оплачивают.
Окончание...
«Выход из Африки»: те же грабли, только в разрезе
Попгенетика и ДНК-генеалогия
К вынесенной в заглавие теме приходится возвращаться снова и снова, но мне не в тягость. Во-первых, тема интересна с точки зрения методологии современной науки, в данном случае популяционной генетики, перенесенной на почву исследования генома. Техническая сложность резко повысилась, но попгенетики остались теми же, при вопиющем отсутствии у них адекватной научной школы. ©По теме: Невероятные неандертальцы

Neandertal Museum in Mettmann, Germany
Во-вторых, на тему геномных исследований практически нет научно-популярных статей, отчасти потому, что популяционные генетики обыкновенно косноязычны, статьи они пишут по стилю и способу изложения ужасные, и научно-популярные статьи у них получаются как на том заводе, который что ни делает, а получается танк. Иначе говоря, у них получается не научно-популярная статья, а такое же вязкое, неудобоваримое «изложение».
Наконец, они не могут писать доходчивых научно-популярных статей по результатам изучения генома человека, потому что они сами не понимают смысл того, что делают. Они берут сотни тысяч и миллионы геномных снипов, что-то там складывают и вычитают по неким прописям, удаляют то, что им по разным причинам не нравится, причем удаления достигают до трети исходных снипов, потом закладывают в компьютер, тот пережевывает и что-то выплевывает, они это «интерпретируют», причем так, что волосы дыбом становятся, и результат публикуют как якобы научное исследование.
Но это еще полбеды, а самая беда у попгенетиков от генома имеет ментально фундаментальный характер. Главный критерий у них - «похожесть». Вот эту «похожесть» они оформляют в самых разных видах. Основной - это «метод главных компонент». Вся его суть - это найти «похожесть» между сравниваемыми величинами, которые берутся самые разные. Если немного утрировать, но только немного - они берут геномы некого индивидуума и его дяди, то есть брата отца. Геномы оказываются похожими, что неудивительно. Незамедлительно следует вывод, что этот индивидуум произошел напрямую от дяди. Или, напротив, дядя - от индивидуума. Это уже как они порешат. Это и называется «интерпретация». Примеров в академических журналах - масса…
Только недавно мы разбирали статью, в которой нашли, что геном ископаемых ямников (это - древние обитатели ямной культуры) похожи на геном древней культуры шнуровой керамики. Вывод - люди шнуровой керамики произошли от ямников, и получили от последних индоевропейские языки. То, что у ямников гаплогруппа R1b, а в шнуровой керамике - гаплогруппа R1a, на это они внимания не обратили. Гаплогрупп в статье они вообще не разбирали. Видимо, посчитали лишним - геном же похож, не так ли? То, что R1a и R1b - это «братья», которые произошли от одного «родителя» - гаплогруппы R1, и поэтому геном у них обязан быть похож - об этом, видимо, не подумали. На гаплотипы и не посмотрели, да их и не определяли. А гаплотипы сразу бы показали, что между R1a и R1b - не менее 40 тысяч лет разницы, да и между гаплотипами ямников и европейцев, древних или современных - тоже тысячелетия разницы. Не могли носители гаплогруппы R1b, эрбины, пройти напрямую из ямной культуры в Европу, во всяком случае, те ямники, геномы которых изучали.
Наконец, те субклады гаплогруппы R1b, которые были у тех ямников, а именно в основном R1b-L23-Z2103, в современной Европе почти не наблюдаются, их нашли у всего нескольких европейцев из десятков тысяч носителей гаплогруппы R1b в Европе, причем почти все европейцы имеют субклады R1b-U106 и R1b-P312 и нисходящие от них, которые не произошли от ямных R1b-L23-Z2103, это совсем другие ДНК-линии. Иначе говоря, не прошли те ямники и их потомки в Европу. Они, в лучшем случае, прошли на юг, на Кавказ и в Месопотамию, а в худшем (для них) случае - просто вымерли. Так что не принесли они индоевропейские языки в Европу, да и не могли принести. И эта грубая ошибка геномных попгенетиков произошла только потому, что не знают они ДНК-генеалогии, не хотят рассматривать.
Это просто один пример из десятков, когда «похожесть» геномов прямолинейно и бестолково берется как главный критерий. Ситуация напоминает ту, которая была в археологии в середине и второй половине прошлого, 20-го века, когда такой же критерий «похожести» показал свою несостоятельность, и в итоге археологи надолго отказались от изучения древних миграций, когда изучение основывалось на «похожести». Они поняли, что не могут отделить миграции от «культурной диффузии», и не могут определить направления миграций, когда основывались на «похожести» - то ли отсюда туда, то ли оттуда сюда. Тогда же был выдвинут лозунг «горшки - не люди», в том смысле, что похожие горшки в двух разных отдаленных местах не означает, что создатели этих горшков передвигались вслед за ними. Так что пора геномным попгенетикам, наконец, сообразить, что похожесть геномов не указывает на направления миграции древних людей, и перестать наступать на те же грабли.
Технически современная генетика вышла на такие рубежи, о которых нельзя было и помыслить еще несколько лет назад. Стало возможным выявлять в ДНК любого человека миллионы снипов, то есть практически необратимых мутаций, которые составляют уникальную мозаику, то есть картину снипов каждого. Эта мозаика отражает миллионы лет эволюции человека, и в эту картину входит множество снипов, унаследованных от общих предков человека и древних предков макаки, орангутанга, гориллы, шимпанзе. Эти снипы затем ушли к неандертальцам (у нас с неандертальцем общих снипов не менее 99%), древним африканцам, древним неафриканцам и их потомкам, и они есть в ДНК любого из нас, миллионы этих древних снипов.
Ну, и что с этим добром делать? Техника - в буквальном смысле на грани фантастики - нам это предоставила, и, как это бывает, обогнала мыслительный аппарат человека. Мыслительный аппарат эти миллионы снипов не схватывает, вручную их обработать нельзя. К счастью, компьютерная обработка данных тоже вышла на уровень граней фантастики, как и способы хранения и переработки информации. Осталось все это совместить.
И вот здесь начинаются большие проблемы. Человек ведь мыслит линейно, за редким исключением людей очень одаренных, которых называют гениями. Поэтому ординарные люди, исследователи генома, стали, естественно, применять к столь сложной проблеме простые линейные подходы, рассуждения, размышления. Выявил компьютер, что у американских индейцев усредненный геном на 11% схож с геномом усредненного русского, вот исследователь и записал: (1) индейцы с русскими совпадают по геному на 11%, значит, (2) русские произошли от американских индейцев. Причем, заметим, что это не компьютер говорит, это исследователь так трактует данные. Как он, исследователь, захочет. Причем, заметьте, не американских индейцев выводит из русских, а наоборот, хотя 11% похожести можно трактовать в любую сторону. На самом деле там похожесть вообще не имеет отношения к поставленному вопросу. У самого исследователя геном на 95+% схож с геномом шимпанзе, видимо, можно с таким же успехом записать, что шимпанзе произошел от того же исследователя. Разберем это чуть подробнее, нам это понадобится для дальнейшего изложения.
История с шимпанзе
Начнем с того, что компьютер сам не скажет, что геномы русского и американского индейца совпадают на 11%, или на сколько там еще. Для этого исследователь закладывает в компьютер программу и исходные условия, определения и допущения. Что там «совпало на 11%»? Там ведь миллионы снипов, включая снипы от общих предков с шимпанзе и прочих приматов, и совпадение с ними уже больше, чем на 90%. Иначе говоря, 11% - это уже означает, что большинство снипов «отфильтрованы», причем фильтрование тоже производится на условиях и по правилам, закладываемых исследователями. Это они диктуют компьютеру, как надо сортировать и фильтровать данные. При этом надо в первую очередь отфильтровать общие снипы древнего общего предка человека и шимпанзе. А его генома у нас нет. Поэтому генетики берут его «прокси», то есть представителя-заместителя, а именно, геном шимпанзе из современного зоопарка. Но наш шимпанзе - неважный прокси, так как его отделяет от того общего предка около пяти миллионов лет. За это время новых снипов у нашего шимпанзе образовалась масса, в абсолютном исчислении. Можно даже подсчитать, сколько. При скорости мутации 0.7х10-9 на пару нуклеоидных оснований в год, за 5 миллионов лет набежит в Y-хромосоме с ее 58 миллионов пар оснований 0.7х10-9 х 5х106 х 58х106 = 203 тысячи мутаций.
Часто работают с фрагментом Y-хромосомы размером в 10 миллионов пар оснований, но и там набежит 35 тысяч «лишних» мутаций, которые есть у нашего шимпанзе, но которых не было 5 миллионов лет назад у его общего с человеком предка. Иначе говоря, когда «фильтруют» снипы, то вычитают 203 тысячи лишних мутаций в расчете на Y-хромосому. Но какие лишние - неизвестно, потому что исходных данных давностью 5 миллионов лет назад нет.
Хуже то, что и шимпанзе разные, так что с мутациями опять неясность. Вот - простой пример. Работая над публикацией про гаплотипы древнего предка человека и шимпанзе, мы с коллегами провели поиск в геномной базе данных (European Nucleotide Archive и National Center for Biotechnology Information (NCBI) GenBank) по основным маркерам шимпанзе из панели наиболее «медленных» маркеров Y-хромосомы, принятых для человека, и получили следующие аллели, то есть в данном случае количество тандемных повторов (DYS 426, 388, 392, 455, 438, 578, 641, 472, 425, 594, 436, 490, 617, 568, 640, 492).
8 15 10 4 5 9 10 5 10 4 4 7 4 4 8 9
Это и есть гаплотип современного шимпанзе, во всяком случае, того, геном которого был заложен в базы данных, указанные выше. Как показали расчеты по этим маркерам, общий предок современного человека и шимпанзе жил 4.0±1.4 миллионов лет назад, что совершенно разумно, и согласуется с антропологическими датировками.
Через некоторое время в базе данных YSearch под номером 6RCUU появился гаплотип шимпанзе, представленный туда известным генетиком Томасом Краном, из источника, который описан так: Chimpanzee cell line Coriell NA03448. Гаплотип оказался совсем другим, хотя аллель при одном маркере - DYS641 - совпадала, была равна 10. Маркеры в основном тоже были другими (DYS393, 426, 388, 389-1, 458, 437, GATA H4, 570, 442, 578, 537, 641, 511, 444, 520, 446, 640, 492, 461, GATA A10, GAAT1B07, 441, 485, 533, 717), причем удивительно, что Кран использовал столь «быстрые» маркеры, ведь применять их к шимпанзе большого смысла не имеет, если сравнивать с маркерами человека. Впрочем, возможно, что у Крана были другие задачи. Но сейчас для нас главное не задачи Крана, а то, что на медленных маркерах общий предок современного человека и шимпанзе (по данным Крана) жил 3.8±1.5 миллионов лет назад, что тоже вполне разумно. Иначе говоря, шимпанзе Крана, как и «наш» шимпанзе, вполне «легитимны», но совершенно различны по гаплотипам. Гаплотип шимпанзе Крана был следующий:
10 10 12 12 15 14 9 4 14 5 9 10 13 10 18 21 12 10 13 5 7 9 14 9 19
и на самых медленных маркерах был совсем другой по сравнению с «нашим» шимпанзе:
10 12 5 10 12 10 (шимпанзе Крана)
8 15 9 10 8 9 (наш шимпанзе)
(маркеры DYS 426, 388, 578, 641, 640, 492).
И вот теперь главный вопрос - геном какого шимпанзе вычитали геномные попгенетики? Проверяли ли они на вычитание другого шимпанзе? Зная попгенетиков, ответ совершенно ясен - конечно, не проверяли. Как получилось, так и получилось. Но тогда какую научную ценность имеет то, что осталось после вычитания? Вопрос, впрочем, риторический. Кого бы там ни вычитали, все равно вычитали не тот набор мутаций, и в остатке после вычитания оказываются как сотни тысяч лишних снипов (которых не оказалось в данном конкретном шимпанзе в зоопарке), так и сотни тысяч снипов недостающих (которые были избыточно вычтены).
И вот сравнивают геномные попгенетики этот перекошенный геном после избыточного или недостаточного вычитания, скажем, с геномом неандертальца (точнее, неандерталки, все расшифрованные геномы неандертальцев, во всяком случае до последнего времени, принадлежали самкам), и говорят - о, смотрите, в них есть мутации, которых не было у шимпанзе (у того шимпанзе, из зоопарка!), но есть у современных людей. Значит, в нас есть доля неандертальцев! Мы произошли от неандертальцев! Пресса, конечно, сходит с ума от экстаза, бросает в воздух чепчики, не понимая, и не желая понимать и задавать вопросы (а зачем это им - сенсация ведь), что это не доля неандертальца, а просто не то вычитали. Из генома неандертальца ведь опять же того шимпанзе вычитали, из зоопарка, и опять сальдо-бульдо получилось дефектным. Причем поскольку тот, из зоопарка, был тем же, «референсным», то недостача или избыток снипов как у человека, так и у неандертальца были симметричными, одними и теми же. Вот и оказывается, что у человека тот же избыток снипов, что и у неандертальца. Сколько? Да те же 3%, которые находят по всему миру, кроме африканцев. Поскольку они разошлись с неафриканцами примерно 160 тысяч лет назад, то у них в определенной степени набежали одни снипы и не набежали другие, вот и разница.
И тогда станет понятно, почему исследования Крингса в 1997 году, которые тогда считали очень важными, а теперь и не упоминают, и которые показали, что мтДНК у современных людей и у неандертальцев принципиально расходятся (см. рис. ниже)

Среднее различие в последовательностях мтДНК между различными людьми на планете (первая диаграмма, среднее различие в 8±3 нуклеотидов), между людьми и неандертальцами (вторая диаграмма, среднее различие в 27±2 нуклеотидов), и людей и шимпанзе (третья диаграмма, среднее различие в 55 нуклеотидов). Такие же результаты были найдены и с другим неандертальцем, найденным в 2500 километрах от первого.
Вот так и получается - по мтДНК никакого близкого сходства между современными людьми и неандертальцами нет, по Y-ДНК не изучали (потому что все изученные по ДНК неандертальцы, повторяю, самки), но невероятно, что гаплотипы современных людей будут похожи на неандертальские, а геном при всех упомянутых манипуляциях оказался схож на 3%. И никто не приводит погрешности этих трех процентов, школа такая у попгенетиков, точнее, ее отсутствие. Что, если там 3±15%, что, скорее всего, и есть?
А то, что у африканцев этих трех процентов нет, как уверяют нас геномные исследователи, в то время как у неафриканцев есть, как-то ломает «теорию выхода» всех людей из Африки. Потому что тогда у всех людей, черных и белых, должны быть те самые 3% генома неандертальцев, куда же он делся? Остается вспомнить известный анекдот про трусы и крестик. Правда, и там нашли какое-то неуклюжее объяснение, не будут же попгенетики из-за каких-то неандертальцев терять столь ценную теорию выхода из Африки…
Конечно, я излагаю здесь картину в качественном виде, чтобы показать, в чем фундаментальные проблемы у попгенетиков с геномным анализом. Они сейчас, конечно, начнут выискивать у меня несущественные мелочи, типа что их «референсный» шимпанзе был не из зоопарка, а из Национального парка, только это сути не меняет. У него в любом случае около пяти миллионов эволюционной разницы с тем, геном кого надо было вычитать. Другие скажут - критиковать легко, а что вы сами предлагаете? Я отвечу, что предлагаю честно рассматривать и описывать данные в научных публикациях, и честно же сообщать, где данные шаткие или просто неверные, а не делать вид, что все правильно и других вариантов нет. Далее, я предлагаю пересчитывать данные не с одним шимпанзе, а с несколькими, поскольку геном получен не только для одного. Тогда станет ясно, что «доля неандертальца» в нас не те 3%, о которых постоянно докладывают, а может оказаться просто нулевой. И тогда надо честно же признать, что ошибка вышла, не подумали, статьи придется отзывать. Да, неприятно, зато честно по отношению к науке и читателям, которые в конечном итоге те геномные исследования и оплачивают.
Окончание...